Pythonでは基本的に、Global Interpreter Lockといって、一つのプロセスでしか動かせない、という制約がある。これは、複数スレッドを立ち上げてPythonコードを実行しようとしても、CPUでの計算を必要とする処理は、一つのスレッドでのみ実行され、他のスレッドは待機状態になる、という制約になる。つまり、CPUを待つ以外の、DBからの返答待ちなどには、PythonのGlobal Interpreter Lock下でも、マルチスレッドによる並列化が意味を成すのだが、CPUバウンドな処理を並列化しようとすると、Pythonではできない。
ただ、できないといっても、Pythonのプロセスを複数立ち上げれば、CPUバウンドな処理でも実用的な並列化が可能になる。コンソールから複数のPythonスクリプトを実行してもいいし、Pythonの標準ライブラリのmultiprocessingを使うともっと簡単にマルチプロセスによる並列化を実現できる。ここで、よく知られるTipsではあるが、マシンのCPU数を超えるプロセスを同時に実行しようとすると、CPU資源の奪い合いによりパフォーマンスが低下する。ちなみに、よく4コア8スレッドというような記載のされ方をするが、これは、物理コア数4、論理コア数8という意味で、ハイパースレッディングと呼ばれる技術のおかげで、1つの物理コアで2つのプログラムを、互いのCPU以外を待つ時間の無駄を利用して交互に実行する仕組みで、Pythonの並列計算ライブラリに並列数を指定するときは、典型的にはcpu_numというような引数だったりするのだが、論理コア数を指定するのがよい。ハードウェアレベルで無駄のないように実行してくれるので、逆に物理コア数を指定すると、CPUの利用率が下がる。
また、NumpyやScipyのような計算速度が重要になりやすいライブラリでは、最初から複数スレッドでマルチコアを活用できるようになっている。これは、これらのライブラリが内部がC言語などのGlobal Interpreter Lockの影響を受けない言語で書かれているためである。これらのライブラリを使うと、何も意識せずに、マルチコアの恩恵を受けられる。
さて、今回のお題は、Pythonでマルチプロセス処理をするときに、各プロセスで実行するPythonコードの中にNumpyなどの複数CPUを活用するライブラリを使ったコードが含まれると考慮する必要がある、Over Subscriptionという問題である。この問題は、例えば12CPUを持つマシンで、12プロセスを立ち上げ、それぞれで12CPUまで活用するNumpyコードを実行したときに、CPU資源の奪い合いが起こり、パフォーマンスが低下する問題である。この問題を解決するための、最も有効な方法は、各プロセスが活用できるCPU数を1にすることである。これは、Numpy、Scipyなど、個々のライブラリの挙動を制御しなくてはならないので、一見面倒に思えるものの、実際にはいくつかの環境変数を指定することで、包括的に制御できることが多い。というのも、並列計算が可能なライブラリのベースにはIntel Math Kernel Library (Intel MKL)や、OpenBLAS、OpenMPなどの数学ライブラリ、並列化ライブラリがあることが多く、これの動きを制御できてしまえば、その上に載るPythonライブラリの挙動も制御できることが多いためである。
例えば、以下のようにPythonコード内で指示すれば、多くのライブラリで並列数が1になる。
import os
os.environ["OMP_NUM_THREADS"] = "1"
os.environ["OPENBLAS_NUM_THREADS"] = "1"
os.environ["MKL_NUM_THREADS"] = "1"
os.environ["VECLIB_MAXIMUM_THREADS"] = "1"
os.environ["NUMEXPR_NUM_THREADS"] = "1"
また、世の中には便利なライブラリがあるもので、threadpoolctlというライブラリを使えば、以下のように書くことで、with内のコードを並列数1で実行できる。
import numpy as np
from threadpoolctl import threadpool_limits, threadpool_info
with threadpool_limits(limits=limits, user_api='blas'):
a = np.random.rand(1024, 1024)
for i in range(128):
b = a @ a
以上、前置きのようなところになるが、実際にOver Subscriptionによって、パフォーマンスが低下するか調べてみた。ベンチマークというには簡単な測定なので悪しからず。CPUはCore i7 5820K (6コア 12スレッド)であり、プロセス当たり並列数が1の場合、12プロセスまではパフォーマンスの低下なく実行できる。CPUの温度をモニタリングしながら、サーマルスロットリング(計算負荷が上がってCPU温度が上昇しすぎた時に、システムを保護するために計算量を抑える仕組み)が発生しない条件下で測定を行った。ついでに、NumpyはOpenBLASがデフォルトで使われるが、MKLを使うように設定すると速くなるという話があるので、それの比較もしてみた。
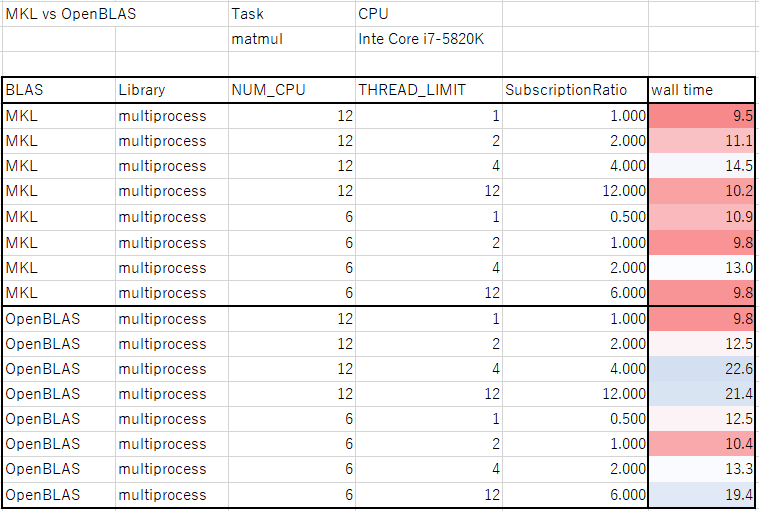
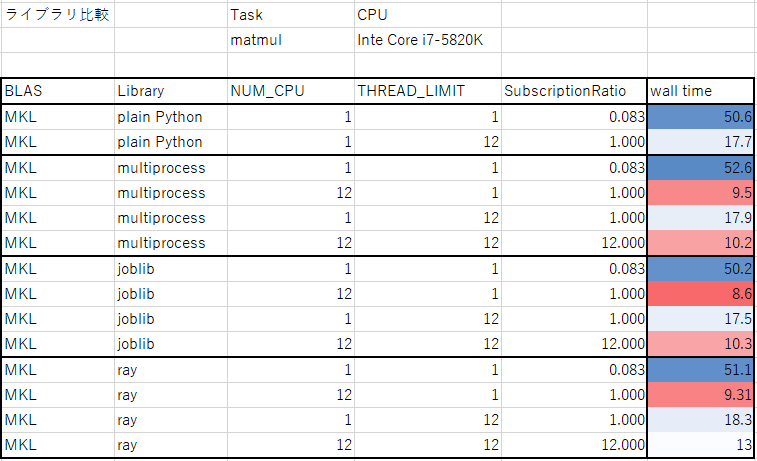
さらに、並列計算のためのライブラリの比較も行った。ライブラリによって、オーバーヘッドが異なる。オーバーヘッドは、ライブラリの機能が増えると大きくなる傾向があるので、必要な機能に応じて選ぶのがよさそう。
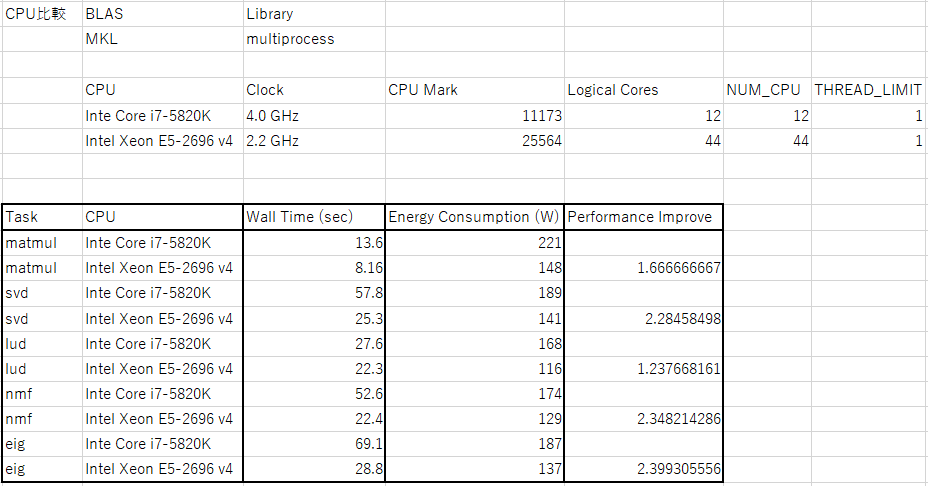
最後に、ちょうどCPUを変更したところだったので、比較を行った。もう一つのCPUはXeon E5-2696v4 (22コア 44スレッド)であり、プロセス当たり並列数が1の場合、44プロセスまではパフォーマンスの低下なく実行できる。主な関心は、CPUMarkのスコアとNumpyの計算速度がどれくらい相関があるのかということだった。CPUMarkのスコアはXeonがi7の2.28倍であるのに対して、Numpyの計算速度の倍率はその周辺でばらついている。ベンチマークは特定の計算でのパフォーマンスを測定するのみであるので、自分で使う計算で実際にパフォーマンスを測定することが重要であることが再確認できた。
測定に使ったコードは以下の通り(Xeonで使ったものなので、並列数44)。
N = 192 * 2
from multiprocess import Pool
# 行列の積
def func_matmul(limits):
import numpy as np
from threadpoolctl import threadpool_limits
with threadpool_limits(limits=limits, user_api='blas'):
a = np.random.rand(512, 512)
for i in range(32):
b = a @ a
return
p = Pool(44)
ret = p.map(func_matmul, [1 for _ in range(N)])
# 特異値分解
def func_svd(limits):
import numpy as np
from threadpoolctl import threadpool_limits
with threadpool_limits(limits=limits, user_api='blas'):
A = np.random.rand(256, 512)
for i in range(32):
U, s, Vh = np.linalg.svd(A)
return
p = Pool(44)
ret = p.map(func_svd, [1 for _ in range(N)])
# LU分解
def func_lud(limits):
import numpy as np
import scipy.linalg
from threadpoolctl import threadpool_limits
with threadpool_limits(limits=limits, user_api='blas'):
A = np.random.rand(512, 512)
b = np.random.rand(512)
for i in range(32):
(LU,piv) = scipy.linalg.lu_factor(A)
P, L, U = scipy.linalg.lu(A)
return
p = Pool(44)
ret = p.map(func_lud, [1 for _ in range(N)])
# 非負値行列因子分解
def func_nmf(limits):
import numpy as np
from sklearn.decomposition import NMF
from threadpoolctl import threadpool_limits
A = np.random.rand(128, 128)
model = NMF(n_components=16, init='random', random_state=0)
with threadpool_limits(limits=limits, user_api='blas'):
for i in range(32):
W = model.fit_transform(A)
return
p = Pool(44)
ret = p.map(func_nmf, [1 for _ in range(N)])
# 固有値分解
def func_eig(limits):
import numpy as np
from threadpoolctl import threadpool_limits
A = np.random.rand(256, 256)
with threadpool_limits(limits=limits, user_api='blas'):
for i in range(32):
a = np.linalg.eig(A)
return
p = Pool(44)
ret = p.map(func_eig, [1 for _ in range(N)])