今回はPythonで細胞数のカウントをしてみます。C++での粒子カウントを解説したものはこちらになります。
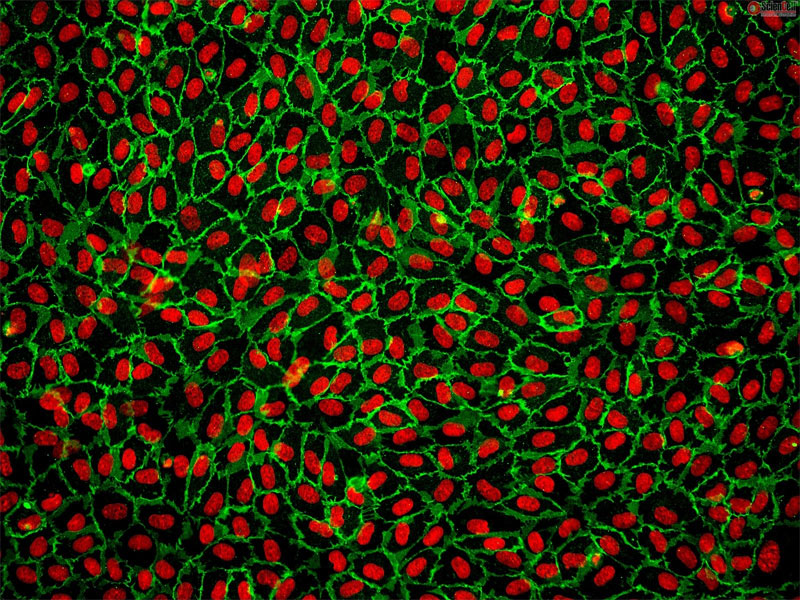
ヒト臍帯静脈内皮細胞 (HUVEC) CD31 の免疫蛍光染色 200× コスモバイオ
画像のチャンネル分離
cv2.imread(‘cell.jpg’ ,1)の最後の1はカラーで読み込むことを表します。ここを0にするとグレースケールで読み込まれます。元画像は3チャンネル(BGR)の画像なので、チャンネルを分離します。OpenCVのsplit関数を使います。NumPyでは3チャンネルがBGRの順に並ぶため、例えば、今回は核の情報を元にカウントを行いますので、赤を表示する時には012のうちの2を指定します。
import cv2
import numpy as np
import matplotlib.pyplot as plt
img = cv2.imread('cell.jpg' ,1)
img_chs = cv2.split(img)
plt.imshow(img_chs[2]) #赤を表示
plt.colorbar()
plt.show()

2値化する
赤のチャンネルを元に、2値化を行います。閾値を100にして、2値化を行いますが、今回はOpenCVの関数を使わずにNumPyの機能で2値化を行います。binimg = (img_chs[2]>100) とすることで、条件を満たすピクセルのみが1になった2値画像が得られます。
import cv2
import numpy as np
import matplotlib.pyplot as plt
img = cv2.imread('cell.jpg' ,1)
img_chs = cv2.split(img)
binimg = (img_chs[2]>100)
plt.imshow(binimg)
plt.colorbar()
plt.show()

距離マップ(distance map)を計算する
次に核の中心部分の座標を求めるために、distance mapを計算します。distanceTransform関数は決まった型のNumPy配列しか受け取らないので、binimg.astype(np.uint8)というようにnp.uint8型に変換しています。uint8というのはunsigned int 8bitを意味しており、符号なし8ビット整数、すなわち、0~255の整数を保持する型になります。
import cv2
import numpy as np
import matplotlib.pyplot as plt
img = cv2.imread('cell.jpg' ,1)
img_chs = cv2.split(img)
binimg = (img_chs[2]>100)
binimg = binimg.astype(np.uint8)
distmap = cv2.distanceTransform(binimg,1,3)
plt.imshow(distmap)
plt.colorbar()
plt.show()

わかりにくいので、一部のみを拡大して表示します。下のコードは0<x<100,0<y<100の正方形領域を抜き出して描画しています。
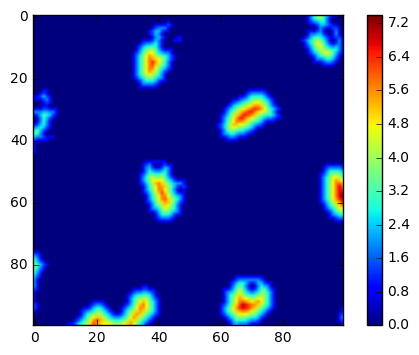
このように、核の輪郭から遠い部分で高い値をもつ画像が得られることになります。
plt.imshow(distmap[0:100,0:100])
plt.colorbar()
plt.show()
距離マップの極大値をもつ場所を計算する
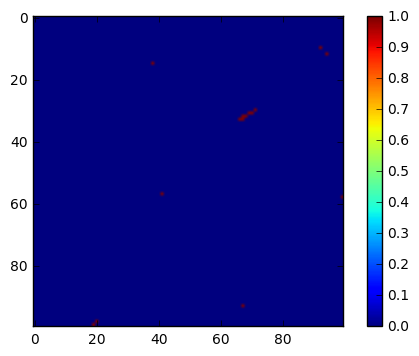
極大の計算に関数を用いてもいいのですが、今回は簡単に、「極大=周辺の20*20ピクセルの領域のどこよりも値が大きい場所」ということにして、計算してみます。
import cv2
import numpy as np
import matplotlib.pyplot as plt
img = cv2.imread('cell.jpg' ,1)
img_chs = cv2.split(img)
binimg = (img_chs[2]>100)
binimg = binimg.astype(np.uint8)
distmap = cv2.distanceTransform(binimg,1,3)
out = distmap*0
ksize=10
for x in range(ksize,distmap.shape[0]-ksize*2):
for y in range(ksize,distmap.shape[1]-ksize*2):
if distmap[x,y]>0 and distmap[x,y]==np.max(distmap[x-ksize:x+ksize,y-ksize:y+ksize]):
out[x,y]=1
plt.imshow(out[0:100,0:100])
plt.colorbar()
plt.show()
膨張させ、輪郭検出し、数える
ここまでで極大値をもつピクセルを検出してきましたが、ちょうど同じ値のピクセルが近くにある可能性は否定できません。そこで、dilate関数によって膨張処理を施し、もし近くに同じ極大値をもつピクセルがあった場合には1つの塊としてまとめてしまいます。dilate関数は第二引数に、カーネルの大きさを指定します。今回は3*3の大きさのカーネルを指定しており、値が0でない各点は3*3マスの大きさに膨張します。膨張処理の後、findContours関数によって輪郭検出を行います。この関数は決まった型のNumPy配列しか受け取らないので、out.astype(np.uint8)というようにnp.uint8型に変換しています。輪郭検出の後は、各輪郭について、輪郭構成点の重心を求め、核の座標としてリストarrに追加しています。arr = np.array(arr)によってリストをNumPy配列に変換していますが、今回は特に必ずすべきという処理ではありません。
import cv2
import numpy as np
import matplotlib.pyplot as plt
img = cv2.imread('cell.jpg' ,1)
img_chs = cv2.split(img)
binimg = (img_chs[2]>100)
binimg = binimg.astype(np.uint8)
distmap = cv2.distanceTransform(binimg,1,3)
out = distmap*0
ksize=10
for x in range(ksize,distmap.shape[0]-ksize*2):
for y in range(ksize,distmap.shape[1]-ksize*2):
if distmap[x,y]>0 and distmap[x,y]==np.max(distmap[x-ksize:x+ksize,y-ksize:y+ksize]):
out[x,y]=1
out = cv2.dilate(out,(3,3))
contours, _ = cv2.findContours(out.astype(np.uint8), cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_NONE)
arr=[]
for i in contours:
x_=0
y_=0
for j in i:
x_ += j[0][0]
y_ += j[0][1]
arr.append([x_/len(i), y_/len(i)])
arr = np.array(arr)
plt.imshow(out[0:100,0:100])
plt.colorbar()
plt.show()
print len(arr)
print arr
399
[[267 577]
[144 578]
[705 575]
[ 25 574]
:
:
[354 12]
[ 92 10]]
と表示されました。
個数は399個で、その座標が続いて順に出力されます。
トラッキングにしても最初は各フレームから対象の座標を取り出さなくてはなりませんので、このような粒子カウントは、画像解析をする上できっと役に立つものだと思っています。